检测snp多态性的方法(SNP检测方法)
1.SNP检测方法有哪些
但不管哪一种方法,首先必须进行靶序列的扩增,然后才能进行其它检测。
传统的SNP检测方法是采用一些已有的成熟技术,如DNA测序、限制性酶切片段长度多态性(RFLP)、单链构象多态性(SSCP)、等位基因特异 的寡聚核苷酸杂交(ASO)等。这些技术虽在某种程度上能完成对SNP的检测,但由于它们必须通过凝胶电泳进行检测,因此,距快速、高效、自动化的目标还 相差甚远。
传统的RFLP只能检测到SNP的一部分,测序技术既费时费力,又不易实现自动化,而且DNA链的二级结构还容易造成人工假相,使测序结果出现 偏差,不适宜于SNP的检测;SSCP则很难满足自动化的需要,难以大规模开展工作。因此,这些方法均未被广泛采用。
DNA芯片技术是近年来新开发的一种DNA序列变异检测工具。DNA芯片(DNA chip),也称生物芯片(biochip),其大小与计算机上的CPU芯片相似,约1 cm2或更大些,以玻璃、硅、聚丙烯等作 为载体基片,芯片上铺了一层肉眼看不见的DNA纤维“地毯”,即具有特定碱基序列的探针。
待测基因经提取后,被切成长短不一的片段,经荧光化学物质标记 后,注射到嵌有芯片的载片上。
2.什么是SNP SNP怎么检测
SNP,即单核苷酸多态性,这种人类最常见的可遗传变异占据了所有已知多态性的90%以上,因此作为研究个体对不同疾病的易感性或者个体对特定药物的不同反应有着重要的意义,也因此国际人类基因研究学会花费大量的资金来完成人类单体型图谱。
SNP分析从根本上来说其实就是确定一对染色体的每种基因的两个拷贝,哪种点突变在这两个拷贝中存在,因此利用定量PCR方法,并配合芯片技术可以快速灵敏的检测到SNP结果。ABI号称有数以千记的定量PCR试剂盒,自然在这方面也不会留下空白,TaqMan SNP Genotyping Assays包含了一百万的HapMap SNPs(人类单核苷酸多态性图谱SNPs),方便SNP分型高通量研究。
传统的SNP检测方法是采用一些已有的成熟技术,如DNA测序、限制性酶切片段长度多态性(RFLP)、单链构象多态性(SSCP)、等位基因特异 的寡聚核苷酸杂交(ASO)等。这些技术虽在某种程度上能完成对SNP的检测,但由于它们必须通过凝胶电泳进行检测,因此,距快速、高效、自动化的目标还 相差甚远。
传统的RFLP只能检测到SNP的一部分,测序技术既费时费力,又不易实现自动化,而且DNA链的二级结构还容易造成人工假相,使测序结果出现 偏差,不适宜于SNP的检测;SSCP则很难满足自动化的需要,难以大规模开展工作。因此,这些方法均未被广泛采用。
3.snp标记怎样判断有具备多态性
SNP是分布在动物基因组中非常常见的一种分子标记。在人体中每1000bp就可能有一个SNP。需找SNP的目的是为了做连锁分析,在现在先进的技术里,还可以做全基因组关联分析(GWAS)。
然后说说标签SNP,这个涉及到单倍型的问题(对单倍型稍微讲解一下!三个SNP假定是G、C突变,G、T突变和A、T突变,但不是说这些突变在单倍体染色体上里是随意组合的,而是趋向于一种组合方式,比如一条染色体是GGT,另一条是CCA)。因为在基因组中,有些区域是较为紧密连锁在一起的,这个区域可大可小,组成一个单倍型框,在区域里也会存在多多少少的SNP。人们没有必要把这些SNP都找出来,因为它们往往就像是捆绑在一起的。于是从中选出一个代表性的SNP,它可以反映这个区域的情况。然后就方便人们继续做关联分析!继人类基因组计划之后,还做了单倍型的图谱。这也是标签SNP的应用。讲的够清楚吧,呵呵……希望理解,谢谢。
4.基因多态性的主要检测方法有哪些
1.限制性片段长度多态性(Restriction Fragment Length Polymorphism,RFLP):由DNA 的多态性,致使DNA 分子的限制酶切位点及数目发生改变,用限制酶切割基因组时,所产生的片段数目和每个片段的长度就不同,即所谓的限制性片段长度多态性,导致限制片段长度发生改变的酶切位点,又称为多态性位点。最早是用Southern Blot/RFLP方法检测,后来采用聚合酶链反应(PCR)与限制酶酶切相结合的方法。现在多采用PCR-RFLP法进行研究基因的限制性片段长度多态性。
2.单链构象多态性(SSCP):是一种基于单链DNA构象差别的点突变检测方法。相同长度的单链DNA如果顺序不同,甚至单个碱基不同,就会形成不同的构象。在电泳时泳动的速度不同。将PCR产物经变性后,进行单链DNA凝胶电泳时,靶DNA中若发生单个碱基替换等改变时,就会出现泳动变位(mobility shift),多用于鉴定是否存在突变及诊断未知突变。
5.snp标记怎样判断有具备多态性
SNP是分布在动物基因组中非常常见的一种分子标记。
在人体中每1000bp就可能有一个SNP。需找SNP的目的是为了做连锁分析,在现在先进的技术里,还可以做全基因组关联分析(GWAS)。
然后说说标签SNP,这个涉及到单倍型的问题(对单倍型稍微讲解一下!三个SNP假定是G、C突变,G、T突变和A、T突变,但不是说这些突变在单倍体染色体上里是随意组合的,而是趋向于一种组合方式,比如一条染色体是GGT,另一条是CCA)。因为在基因组中,有些区域是较为紧密连锁在一起的,这个区域可大可小,组成一个单倍型框,在区域里也会存在多多少少的SNP。
人们没有必要把这些SNP都找出来,因为它们往往就像是捆绑在一起的。于是从中选出一个代表性的SNP,它可以反映这个区域的情况。
然后就方便人们继续做关联分析!继人类基因组计划之后,还做了单倍型的图谱。这也是标签SNP的应用。
讲的够清楚吧,呵呵……希望理解,谢谢。
6.SNP基因分型的常见方法有哪些
SNP基因分型的常见方法有如下几种:
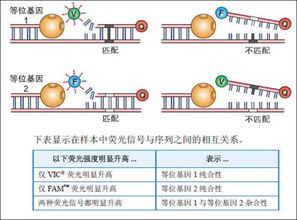
1. TaqMan探针法 针对染色体上的不同SNP位点分别设计PCR引物和TaqMan探针,进行实时荧光PCR扩增。探针的5'-端和3'-端分别标记一个报告荧光基团和一个淬灭荧光基团。当溶液中存在PCR产物时,该探针与模板退火,即产生了适合于核酸外切酶活性的底物,从而将探针5'-端连接的荧光分子从探针上切割下来,破坏两荧光分子间的PRET,发出荧光。通常用于少量SNP位点分析。
2.SNaPshot法 该技术由美国应用生物公司(ABI)开发,是基于荧光标记单碱基延伸原理的分型技术,也称小测序,主要针对中等通量的SNP分型项目。在一个含有测序酶、四种荧光标记ddNTP、紧临多态位点5'-端的不同长度延伸引物和PCR产物模板的反应体系中,引物延伸一个碱基即终止,经ABI测序仪检测后,根据峰的移动位置确定该延伸产物对应的SNP位点,根据峰的颜色可得知掺入的碱基种类,从而确定该样本的基因型。对于PCR产物模板可通过多重PCR反应体系来获得。通常用于10-30个SNP位点分析。
3.HRM法 高分辨率熔解曲线分析(HRM)是近几年兴起的SNP研究工具,它通过实时监测升温过程中双链DNA荧光染料与PCR扩增产物的结合情况,来判断是否存在SNP,而且不同SNP位点、是否是杂合子等都会影响熔解曲线的峰形,因此HRM分析能够有效区分不同SNP位点与不同基因型。这种检测方法不受突变碱基位点与类型的局限,无需序列特异性探针,在PCR结束后直接运行高分辨率熔解,即可完成对样品基因型的分析。该方法无需设计探针,操作简便、快速,成本低,结果准确,并且实现了真正的闭管操作。
4.Mass Array法 MassARRAY分子量阵列技术是Sequenom公司推出的世界上领先的基因分析工具,通过引物延伸或切割反应与灵敏、可靠的MALDI-TOF-MS技术相结合,实现基因分型检测。基于MassARRAY平台的iPLEX GOLD技术可以设计最高达40重的PCR反应和基因型检测,实验设计灵活,分型结果准确性高。根据应用需要,对数十到数百个SNP位点进行数百至数千份样本检测时,MassARRAY具有最佳的性价比,特别适合于对全基因组研究发现的结果进行验证,或者是有限数量的研究位点已经确定的情况。
5.Illumina BeadXpress法 采用Illumina公司的BeadXpress系统进行批量SNP位点检测,可以同时检测1-384个SNP位点,往往用于基因组芯片结果确认,适合高通量检测。微珠芯片具有高密度、高重复性、高灵敏度、低上样量、定制灵活等特点,极高的集成密度,从而获得极高的检测筛选速度,在高通量筛选时可显著降低成本。
声明:本网站尊重并保护知识产权,根据《信息网络传播权保护条例》,如果我们转载的作品侵犯了您的权利,请在一个月内通知我们,我们会及时删除。
蜀ICP备2020033479号-4 Copyright © 2016 学习鸟. 页面生成时间:3.206秒









